青岛腾龙微波科技有限公司
您身边的实验室专家

更新时间:2021-02-20 12:08:02
稳定性同位素标记的糖代谢研究
Ron Orlando
复杂碳水化合物研究中心,生物化学和分子生物学系,化学系,乔治亚大学,雅典,GA 30602 USA
“我的实验室已经使用了一系列的CIL产品,他们的产品在蛋白组学和糖组学定性定量的表现很满意。
CIL的销售人员非常乐于助人并且知识渊博,他们多次在实验设计上帮助我们,如果没有他们的帮助我们无法完成这项研究工作。”--– Ron Orlando
糖基化是真核系统中最常见的蛋白质表达修饰之一。据估计60-90%的哺乳动物蛋白是糖基化的,事实上所有膜和分泌蛋白也是糖基化的。糖蛋白在生理代谢中通常起关键作用,例如细胞识别,信号传导,炎症和癌变。鉴于蛋白质糖基化的重要生理作用,众多研究团队致力于特异性聚糖的结构鉴定,表达蛋白的聚糖,以及对这些结构如何变化的详细研究,例如细胞分化或随着肿瘤细胞的发展。所有这些努力发展出了新的研究领域--糖组学。
质谱(MS)已经发展成为一种分析复杂混合物的方法,以其快速、灵敏、准确而广受欢迎。糖组学研究通常包括质谱分析(MS)聚糖的彻底分解产物。虽然对聚糖进行定性这是一个有效方法,但是在MS定量上存在争议。例如,基质效应,由于其他化合物的存在对分析电离物的离子抑制现象,能够在底物浓度不变的情况下影响指定物质的分析结果。其他干扰质谱分析结果的因素包括不同仪器的检出限,以及仪器存在的误差,和样品处理过程中的损失。不同相对定量方法的成功运用取决于如何解决原始数据存在的误差。
同位素标记技术的使用可以弥补这些定量的缺陷,因此在各种“组学”中得到了广泛的应用,其中在蛋白质组学领域应用最广泛。稳定同位素标记的氨基酸在细胞培养(SILAC)方面就是一个很好的例子,该方法完美的将同位素标记与蛋白质结合,以便用质谱来研究蛋白质组学。在SILAC实验中,两种细胞生长在培养基中,其中一个有“轻氨基酸”(自然丰度),另一个是重氨基酸(同位素标记)(例如,未标记和L-赖氨酸·2HCl(13C6,99%)(CIL目录号CLM-2247)和L-精氨酸·HCl(13C6,99%)(CIL目录号CLM2265))。同位素标记的氨基酸代替了细胞培养中使用的天然氨基酸与所有新和成的蛋白质结合。在细胞分裂过程中,每一个特定的氨基酸都被对应的同位素标记氨基酸替代。这种方法的优势是两种细胞裂解后立即混合在一起,因此来自这两种细胞类型的蛋白质在相同的样品处理过程中的消化、纯化和分离步骤的实验条件完全相同。 因为这个原因,SILAC通常被认为是蛋白质组学定量的金标准。
在这篇文章中描述了糖组学的一种体内标记的方法,这种方法类似于SILAC在蛋白质组学领域的应用。这种糖组学方法叫做IDAWG -(谷氨酰胺对氨基糖的同位素检测)-这基于谷氨酰胺侧链是氨基糖核苷酸合成中唯一的氮供体(图一)。因此,将L-谷氨酰胺(酰胺-15N,98%)引入不含谷氨酰胺的培养基中,可产生N15标记的包括乙酰氨基葡萄糖、乙酰半乳糖胺和唾液酸在内的所有氨基糖类。这导致每个含N和O的聚糖、糖脂和胞外基质多糖的质量数增加+1Da。该方法通过培养在未标记和N15标记的谷氨酰胺环境中的小鼠胚胎干细胞蛋白质释放含N聚糖的实验得到了验证。这些实验的成功使我们预测,未来IDAWG技术将有助于细胞培养方面各种比较糖组学的研究。
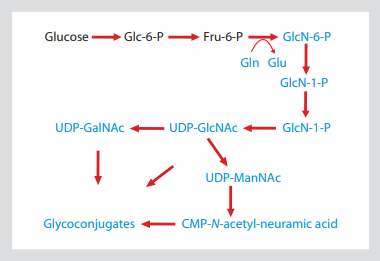
图1:己糖胺生物合成途径表明谷氨酰胺的侧链是糖核苷酸产生过程中氨
基糖的唯一氮供体来源,这允许将15N同位素标记引入所有氨基糖,包括
GlcNAc、GalNAc和唾液酸。含15N的物质用蓝色表示。
结论
己糖胺生物合成途径的第一步也是限速步骤(图1)是糖酵解中间产物的转化(果糖-6-磷酸转化为葡萄糖胺-6-磷酸)。在这个步骤中引入的氨基氮仅由Gln(谷氨酰胺)的侧链酰胺提供,它被转化为Glu(葡萄糖)。葡萄糖胺-6-磷酸是UDP-GlcNAc的前体,UDP-GlcNAc又导致其他主要的含糖核苷酸氨基糖UDPGalNAc和CMP-唾液酸。因此,所有含有GlcNAc-、GalNAc-和唾液酸的分子都是通过在细胞培养基中添加酰胺-15N-Gln来进行同位素标记的靶点。

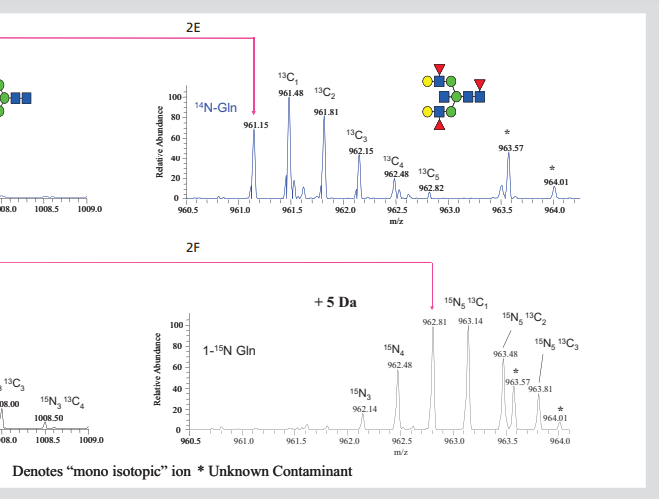
图2。同位素标记的N-连接聚糖。全FT-MS 850-2000m/z二甲基化N-链的光谱在未
标记的Gln中生长的细胞释放的聚糖(2A)或酰胺-15N-Gln(2B)。光谱的扩展区域
显示15N的预期2 Da质量位移进入GlcNAc2Man7的两个核心GlcNAc残基聚糖(2C,
2D)和5-Da增加对应于15N掺入羊膜五糖中GlcNAc5Man3Gal2Fuc3聚糖(2E,2F)。
初步实验,评估使用代谢标记方法将稳定同位素植入培养细胞的聚糖中的可能性。
在这些实验中,R1小鼠胚胎干细胞(mESCs)是在标准条件下培养的。细胞培养基通常不含谷氨酰胺,
因为这种氨基酸在水溶液中分解为谷氨酸和氨。这种方式简化了IDAWG标记方法,因为不需要特别消耗Gln(谷氨酰胺)培养基。使用15N标记的-Gln或未标记的Gln以标准浓度(2 mM)补充培养基。这种直接的替代是唯一改变正常细胞培养程序所需的IDAWG。在这个例子中,小鼠胚胎干细胞用标记或未标记Gln培养三天,然后从蛋白质中分离出N-和O-连接的聚糖,用于质谱分析。通过比较在未标记的Gln或L-谷氨酰胺(酰胺-15N,98%)中生长的细胞释放的过甲基化N-连接聚糖的FT-MS(超高分辨光谱)光谱,研究了15N并入MESC的N-连接聚糖中的情况(图2A和2B)。在这些光谱中观察到的丰富离子对应于单电荷或双电荷的高甘露糖聚糖(GlcNAc2Man5-9)。通过这两种光谱的对比表明,在重介质中生长的细胞获得的聚糖离子,双电荷离子增加了1 m/z单位,单电荷离子增加了2 m/z单位。这个预期的结果,前提是15N已并入这些聚糖中所含的两个核心GlcNAC,并且这是高甘露糖聚糖中唯一的氮。
在仔细观察双电荷分子离子(M+2Na)2+时,可以清楚地看到聚糖质量的这种变化,这种双电荷分子离子是由GlcNAc2Man7聚糖产生的,它分别以1005.5和1006.5 M/z单位出现在14N和酰胺-15N的聚糖中(图2C和2D)。在这些光谱中,响应最强的离子出现在用14N或两个15N计算的单同位素质量处,这表明这种聚糖的大多数都有15N结合到两个可能的位点。存在与标记不足相对应的离子,即零和一个15N的结合;然而响应最强的是完全标记的。
大量的N-连接聚糖通过每个GlcNAc端基插入一个15N而质量相似地增加。这可以通过扩展图2A和2B中所示的光谱来观察,以便仅观察到从960.5到964.1的区域(图2E和2F,分别针对在14N和酰胺15N Gln培养基中生长的细胞释放的聚糖。该区域显示了分子离子,(M+3Na)3+,用于组成GlcNAc5Man3Gal2Fuc3的复杂聚糖。正如所预测的,当从生长在15N-Gln中的细胞中获得该聚糖时,其单同位素离子增加了+5Da。此外,完全标记的聚糖是主要存在形式,然而不完全标记的,却能从标记不完全处观察到离子。
通过计算15N光谱强度的不同同位素,表明该聚糖中含有98%的15N,恰好等于这个实验所用谷氨酰胺中15N的量。对其他聚糖的同位素分布的类似计算表明,它们的15N掺入范围为96-98%。这些实例表明,L-谷氨酰胺(酰胺-15N,98%)中的15N被广泛地并入N-连接聚糖的GlcNAc端基,表明这可能是将稳定同位素引入聚糖的有效方法。
讨论
聚糖的代谢标记为评估在培养中诱导或维持的任何细胞行为过程中聚糖转换的动力学提供了许多新的机会。例如,用L-谷氨酰胺(酰胺-15N,98%)标记细胞,然后用轻Gln代替培养基补充剂,可以测定任何含氨基糖聚糖的半衰期。以前,聚糖转化研究需要结合放射性单糖和广泛的后续分馏,以确定聚糖表达的具体变化。一般来说,这些放射性示踪技术能够非常灵敏的检测聚糖类物质,但在跟踪个别聚糖结构或亚群的生物合成相关物质上分辨率低。本文报道的稳定同位素掺入法将高分辨率质谱的分析优势与理解聚糖转化动力学的生物学必要性结合起来。因此,IDAWG呈现出一种有效的定量方法,探索聚糖、糖蛋白和糖脂在细胞培养系统中的生物学作用。
感谢
本研究由美国国立卫生研究院/NCRR资助的生物医学糖组学综合技术资源(P41 RR018502)和美国国立卫生研究院/NCRR资助的综合糖技术研究资源(P41 RR005351)资助。
SILAC相关产品
|
货号 |
品名 |
货号 |
品名 |
|
CLM-2265 |
L-Arginine·HCl (13C6, 99%) |
DLM-4212 |
|
|
NLM-557 |
L-Glutamine (amide-15N, 98%) |
CLM-653 |
|
|
CLM-2247 |
L-Lysine·2HCl (13C6, 99%) |
CLM-653 |
|
|
DLM-6038 |
CNLM-7821 |
||
|
NLM-395 |
CDNLM-6810 |
||
|
CLM-2051 |
CNLM-291 |
||
|
CLM-1268 |
NLM-1554 |
||
|
CNLM-7819 |
DLM-2641 |
||
|
CDNLM-6801 |
DLM-2640 |
||
|
CNLM-539 |
DLM-2640 |
L-LYSINE:2HCL (4,4,5,5-D4, 96-98%)MICROBIOLOGICAL/PYROGEN TESTED |
|
|
NLM-396 |
CLM-632 |
||
|
NLM-1267 |
CNLM-3454 |
||
|
DLM-541 |
NLM-143 |
||
|
DNLM-7543 |
NLM-143 |
L-LYSINE:2HCL (ALPHA-15N, 98%) MICROBIOLOGICAL/PYROGEN TESTED |
|
|
CLM-2070 |
DLM-570 |
||
|
NLM-395 |
DNLM-7545 |
||
|
ULM-8347 |
NLM-631 |
||
|
CLM-2262 |
ULM-8766 |
||
|
CLM-2262 |
|
|
|
|
CNLM-281 |
|
|
参考
1. Varki, A. 1993. Biological roles of oligosaccharides: all of the theories are correct. Glycobiology, 3(2), 97-130.
2. Krueger, K.E.; Srivastava, S. 2006. Posttranslational protein modifications: current implications for cancer detection, prevention, and therapeutics. Mol Cell Proteomics, 5(10), 1799-1810.
3. Apweiler, R.; Hermjakob, H.; Sharon, N. 1999. On the frequency of protein glycosylation, as deduced from analysis of the SWISS-PROT database. Biochim Biophys Acta, 1473(1), 4-8.
4. Drickamer, K.; Taylor, M.E. 1998. Evolving views of protein glycosylation. Trends Biochem Sci, 23(9), 321-324.
5. Dwek, R.A. 1995. Glycobiology: more functions for oligosaccharides. Science, 269(5228), 1234-1235.
6. Lis, H.; Sharon, N. 1993. Protein glycosylation. Structural and functional aspects. Eur J Biochem, 218(1), 1-27.
7. Haltiwanger, R.S. 2002. Regulation of signal transduction pathways in development by glycosylation. Curr Opin Struct Biol, 12(5), 593-598.
8. Lowe, J.B. 2003. Glycan-dependent leukocyte adhesion and recruitment in inflammation. Curr Opin Cell Biol, 15(5), 531-538.
9. Fukuda, M. 1996. Possible roles of tumor-associated carbohydrate antigens. Cancer Res, 56(10), 2237-2244.
10. Hakomori, S. 1989. Aberrant glycosylation in tumors and tumor-associated carbohydrate antigens. Adv Cancer Res, 52, 257-331.
11. Lowe, J.B.; Marth, J.D. 2003. A genetic approach to mammalian glycan function. Annu Rev Biochem, 72, 643-691.
12. Muramatsu, T. 1993. Carbohydrate signals in metastasis and prognosis of human carcinomas. Glycobiology, 3(4), 291-296.
13. Olden, K. 1993. Adhesion molecules and inhibitors of glycosylation in cancer. Semin Cancer Biol, 4(5), 269-276.
14. Lubner, G.C. 2003. Glycomics: an innovative branch of science. Boll Chim Farm, 142(2), 50.
15. Zaia, J. 2004. Mass spectrometry of oligosaccharides. Mass Spectrom Rev, 23(3), 161-227.
16. North, S.J.; Koles, K.; Hembd, C.; et al. 2006. Glycomic studies of Drosophila melanogaster embryos. Glycoconj J, 23(5), 345-354.
17. Jang-Lee, J.; North, S.J.; Sutton-Smith, et al. 2006. Glycomic profiling of cells and tissues by mass spectrometry: fingerprinting and sequencing methodologies. Methods Enzymol, 415, 59-86.
18. Ong, S.E.; Blagoev, B.; Kratchmarova, I.; et al. 2002. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Mol Cell Proteomics, 1(5), 376-386.
19. Ong, S.E.; Foster, L.J.; Mann, M. 2003. Mass spectrometric-based approaches in quantitative proteomics. Methods, 29(2), 124-130.
20. McClain, D.A. 2002. Hexosamines as mediators of nutrient sensing and regulation in diabetes. J Diabetes Complications, 16(1), 72-80.
21. Yki-Jarvinen, H.; Vogt, C.; Iozzo, P.; et al. 1997. UDP-N-acetylglucosamine transferase and glutamine: fructose 6-phosphate amidotransferase activities in insulin-sensitive tissues. Diabetologia, 40(1), 76-81.
22. Kean, E.L.; Munster-Kuhnel, A.K.; Gerardy-Schahn, R. 2004. CMP-sialic acid synthetase of the nucleus. Biochim Biophys Acta, 1673(1-2), 56-65.
23. Thoden, J.B.; Wohlers, T.M.; Fridovich-Keil, J.L.; et al. 2001. Human UDPgalactose 4-epimerase. Accommodation of UDP-N-acetylglucosamine within the active site. J Biol Chem, 276(18), 15131-15136.
